Researchers in the Center for Artificial Intelligence Research and Department of Bioinformatics, Institute of Medicine, at University of Tsukuba, have constructed a new basic data called the “MOCCS profile” of base sequences bound by transcription factors that control human gene expression. Furthermore, they revealed that transcription factors have specific binding sequences for each type of cell and by applying this profile, they established a method to evaluate the effects of genetic variation on DNA binding of transcription factors.
The characteristics of the diverse cells that make up the human body are manifested by differences in gene expression. This control of gene expression is made possible by transcription factors bound to specific base sequences on the genome. It is difficult to clarify the sequences to which transcription factors bind (transcription factor binding sequences) for each cell type, and which are important for elucidating the control mechanism of each gene expression. Until now, the overall picture of transcription factor binding sequences including commonalities and diversity across transcription factor types and cell types had not been clarified.
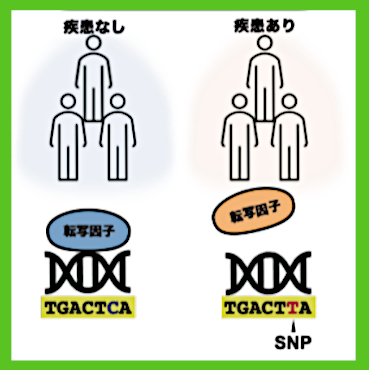
Researchers used large-scale data on binding sites of human transcription factors to construct new basic data on transcription factor binding sequences, “MOCCS profiles,” and analyzed transcription factor binding sequences across transcription factors and cell types. They conducted analysis of the data and the results revealed that approximately half of the transcription factors analyzed had specific binding sequences for each cell type. Furthermore, by applying MOCCS profiles, researchers developed an index that predicted the influence of single nucleotide polymorphisms (SNPs) on DNA binding of transcription factors. It was shown that it was possible to properly assess the impact.
The MOCCS profile constructed during analysis could be combined with epigenomic data, etc, to help understand cell type-specific gene expression control mechanisms and to evaluate the impact of somatic mutations that occurred in cancer cells on the binding of transcription factors. It is expected that the MOCCS profile can be used in many fields. (Translated from “Tsukuba Journal” - Press Release in Japanese Language - University of Tsukuba Website )
→ Publishing Journal - BMC Genomics 【DOI】10.1186/s12864-023-09692-9 "Transcription factor-binding k-mer analysis clarifies the cell type dependency of binding specificities and cis-regulatory SNPs in humans."

ヒトの遺伝子発現を制御する転写因子が結合する塩基配列の基盤データ「MOCCSプロファイル」を新たに構築し、転写因子が細胞の種類ごとに特異的な結合配列を持つことを明らかにしました。また、これを応用し、遺伝的変異が転写因子のDNA結合に与える影響を評価する方法を確立しました。
ヒトの身体を構成する多種多様な細胞の特徴は、遺伝子発現の違いによって現れます。このような遺伝子発現の制御は、ゲノム上で特異的な塩基配列と結合する転写因子によって成り立っており、細胞の種類ごとに転写因子が結合する配列(転写因子結合配列)を明らかにすることは、それぞれの遺伝子発現の制御メカニズムの解明に重要です。しかしながら、これまで、転写因子の種類や細胞の種類に横断的な共通性や多様性といった、転写因子結合配列の全体像は明らかになっていませんでした。
本研究では、大規模なヒト転写因子の結合部位に関するデータを用いて、転写因子結合配列の新たな基盤データ「MOCCSプロファイル」を構築し、転写因子および細胞の種類横断的に、転写因子結合配列の解析を行いました。その結果、解析した約半数の転写因子は、細胞の種類ごとに特異的な結合配列を持つことが明らかとなりました。さらに、MOCCSプロファイルを応用して、一塩基多型(SNP)が転写因子のDNA結合に与える影響を予測する指標を開発し、転写因子・細胞型の観点から疾患関連SNPが転写因子結合に与える影響を適切に評価できることを示しました。
今回構築したMOCCSプロファイルは、エピゲノムのデータ等と組み合わせて、細胞型特異的な遺伝子発現制御メカニズムの理解につなげたり、がん細胞に生じた体細胞変異が転写因子の結合に与える影響度の評価など、多方面での活用が期待されます。(→ プレスリリース 筑波大学ウェブページ)
→ 研究プレスリリース PDF
→ BMC Genomics 【DOI】10.1186/s12864-023-09692-9 "Transcription factor-binding k-mer analysis clarifies the cell type dependency of binding specificities and cis-regulatory SNPs in humans."


